|
Fondazione FONAMA |
fonama@fonama.org |
Tel. 335250742 |
 |
 |
Traduzioni a cura di Natale Marzari
Dopo 41 anni e 5 mesi, nel maggio 2006 la
magistratura di Trento ha riconosciuto l'esistenza e la gravità di quella
malattia rara che nessuna altra istituzione o persona singola della provincia di
Trento ancora mi riconosce, e per negare la quale ancora mi perseguita.
Natale Marzari |
ATPasi F0F1
Questo enzima sostanziale (di circa 500.000 atomi) è facilmente visibile al microscopio elettronico come una sfera di 8,5 nm attaccata sul lato della matrice della membrana mitocondriale
interna. Le sfere possono essere staccate con vari metodi, dopo i quali esse agiscono come ATPasi. La funzione fisiologica di questo enzima è la sintesi dell'ATP, usando l'energia
immagazzinata nel pH transmembranico ed il gradiente di potenziale
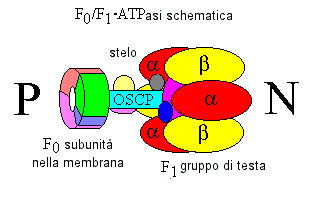
L'insieme completo contiene una catena di 12 tipi diversi di polipeptidi. numerosi di questi sono presenti in molte copie. Il gruppo catalitico di testa è connesso ad uno stelo sensibile
all'oligomicina una piastra di base conduttiva per i protoni nella membrana mitocondriale interna. Per ogni molecola di ATP elaborata dal complesso devono passare tre protoni attraverso la
membrana dalla fase esterna P alla fase interna N del complesso.
Il gruppo di testa F1 contiene tre siti di legame nucleotidico, e l'enzima esegue probabilmente tre cicli-fase catalitici. Nella prima fase, l'ADP ed il fosfato si legano ad un centro
attivo, il quale catalizza il legame ATP. Questo passo è energeticamente possibile poiché l'energia libera rilasciata dal leggero legame dell'ATP nel centro attivo viene compensata
dall'Instabilità del nuovo legame fosfo anidride. L'energia dal protone fornisce la forza motrice richiesta per togliere l'ATP dal centro attivo.
Il pezzo base F0 alloggiato nella membrana mitocondriale interna è una turbina
molecolare mossa dal gradiente protonico transmembranico. L'entrata dei protoni
forza un collod'oca a rotare fra la piastra di base F0 ed il gruppo di testa F1,
alterando la forma della subunità e questo movimento viene effettuato. Una
seconda, delle proteine centrali la quale connette il gruppo di testa con il
piede di base smorza e previene la tensione non necessaria quando lo stelo
centrale ruota. L'energia è trasmessa alle subunità catalitiche nella ATP
sintasi della parte della testa F! con la rotazione del collo d'oca. Il "collo
d'oca" distorce le subunità proteiche, distruggendo la loro capacità di legare
ATP. L'apporto energetico è usato per il rilascio dell'ATP, non per la
formazione del legame.
E' presumibilmente necessario inattivare il meccanismo catalitico nel centro che ha appena formato ATP (per impedire che questo centro idrolizzi i suoi propri
prodotti) prima che distrugga la sua capacità di legare ATP. Questo fa si che il prodotto venga rilasciato. Mentre, gli altri due centri attivi stanno eseguendo la loro parte del ciclo
catalitico. I tre centri attivi operano simultaneamente, ma fuori fase di 120o. Essi fanno si che fino a 9 protoni (con la possibilità di arrivare a 12) producano una rotazione
del collo d'oca e producano 3 molecole di ATP.
Ricorda che l'intero complesso è reversibile. Norfmalmente l'energia del gradiente protonico è usata per fabbricare ATP, ma è egualmente possibile pensare in vitro un'altra
via, ed usare l'idrolisi dell'ATP per ruotare il collo d'oca, fino ad arrivare a pompare protoni all'indietro attraverso la turbina dentro il compartimento extramitocondriale. Se il piede
di base F0 non è attaccato a membrane non sarà utile, ed il complesso farà semplicemente una ATPasi, come originalmente osservato.
E' possibile osservare direttamente la rotazione del collo d'oca della ATPasi F1 usando un microscopio a fluorescenza, sebbene sia necessaria una considerevole ingenuità. Noji et al (1997) Nature 386, 299-302
usarono una ATPasi F1 geneticamente modificata di un batterio termofilo espressa in E. coli. Essi scaricarono il piede di base F0 e fissarono la testa del gruppo motore F1 ad una piastrina
di vetro usando punti di polistidina attaccate al terminale N delle tre sottounità beta. Il vetrino era stato pretrattato con perossidasi horseradish associata al complesso
nichel acido nitrilotriacetico, al quale la polistidina si lega con grande affinità. [L'acido nitrilotriacetico si lega a mezza molecola EDTA, così si ottengono delle posizioni di nichel
scoordinate disponibili per legami esterni.]
I motori sono incollati alle larghe sottounità catalitiche, lasciando gli alberi
motore esposti, e staccati dal vetro. Le unità secondarie gamma che formano
l'albero sono state modificate
sul posto dirigendo la mutagenesi a rimuovere il Cys193
originale (che non conviene lontano dall'albero) e per
sostituirlo con serina. Questi operatori inoltre hanno sostituito Ser107 nella
regione del gambo
con cisteina.
Questo singolo residuo di cisteina (l'unico nella molecola)
potrebbe allora essere biotinilato, e collegato usando un marcatore streptavidin
fluorescente, ai filamenti biotinilati dell'actina. [
Streptavidin ha quattro siti obbligatori della biotina.]
I filamenti fluorescenti dell'actina erano molte volte più
grandi dei motori legati ad essa ed hanno potuto essere visualizzati in un
microscopio ottico.
L'aggiunta di 2mM di ATP ha fatto ruotare in senso antiorario un piccolo
numero di alberi motorio marcati con filamenti
di actina per . Il film mostra i risultati
che hanno ottenuto.
|
Film dell'ATPasi
clicca sulla freccia a sinistra per avviare il filmino
|
Il movimento circolare inoltre si presenta nelle proteine che fanno
ruotare i flagelli batterici, un altro importante sistema di enzimi che è
guidato dall'energia
motrice dei protoni. Appare che la ruota sia in uso continuo da
almeno 2000
milioni di anni.


